Mikroskopia ramanowska to nowoczesna technika, która pozwala zajrzeć w głąb komórek bez ich uszkadzania. W ramach projektu Miesiąc Partnera Klastra, którego Partnerem w sierpniu jest Jagiellońskie Centrum Innowacji zapraszamy do lektury artykułu, w którym odkryjesz, jak ta metoda może śledzić zmiany biochemiczne w komórkach, pomagając w zrozumieniu procesów takich jak apoptoza czy działanie leków. Poznaj fascynujące zastosowania tej technologii w badaniach nad zdrowiem i chorobami!
Spektroskopia ramanowska (RS, z ang. Raman Spectroscopy) jest uniwersalną techniką analityczną wykorzystywaną w wielu obszarach badawczych. Podstawą tej metody jest pomiar energii światła rozproszonego w sposób nieelastyczny.1,2 Dzięki temu, że każda cząsteczka charakteryzuje się inną energią drgań wiązań chemicznych, widmo ramanowskie stanowi chemiczny „odcisk palca”, umożliwiając identyfikację badanych substancji.
Charakterystyka złożonego materiału badawczego wymaga często nie tylko uzyskania informacji chemicznej, ale również określenia dystrybucji zidentyfikowanych składników w próbce. Odpowiedzią na to wyzwanie jest połączenie spektroskopii ramanowskiej z mikroskopią konfokalną, co dało podwaliny pod nową technikę – mikroskopię ramanowską. Wśród dodatkowych zalet takiego rozwiązania wymienić można brak konieczności stosowania znaczników, nieniszczący charakter pomiaru oraz możliwość uzyskiwania widm z próbek w roztworach wodnych. Te cechy sprawiły, że technika ta jest coraz chętniej wykorzystywana do śledzenia procesów komórkowych in vitro czy charakteryzacji chemicznej tkanek.1,3
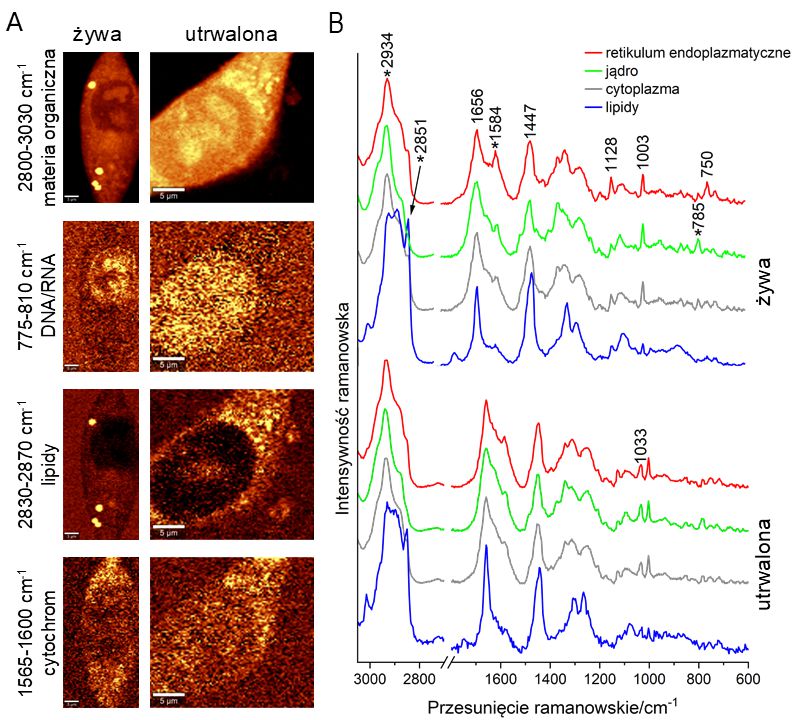
Mikroskopia ramanowska umożliwia uzyskanie informacji o dystrybucji głównych organelli oraz regionów komórki (takich jak jądro, cytoplazma czy obszary bogate w cytochrom) i śledzenie zmian biochemicznych zachodzących w ich obrębie. Dzięki swojej wysokiej czułości i specyficzności może być istotnym narzędziem służącym m.in. do określania stopnia nienasycenia kropel lipidowych czy stanu utlenienia jonu żelaza w cytochromach.4,5 Pozwala to na śledzenie procesów komórkowych, takich jak np. apoptoza6, czy monitorowanie efektów działania substancji biologicznie czynnych oraz ich dystrybucji wewnątrzkomórkowej.
Coraz częściej wykorzystywana jest do identyfikacji komórek bądź tkanek zmienionych chorobowo, dostarczając nowych informacji na temat rozwoju patologii różnego pochodzenia. Technika ta jest jedną z metod wykorzystywanych rutynowo w diagnozie malarii dzięki wysokiej czułości na detekcję hemozoiny – pochodnej hemoglobiny.7 Duże nadzieje wiąże się również z zastosowaniem mikroskopii ramanowskiej do okołooperacyjnego określania granic tkanek zmienionych nowotworowo.8 W poniższym artykule zostanie przedstawiony przykład zastosowania tej techniki do detekcji zmian biochemicznych zachodzących w linii komórkowej mysich fibroblastów tkanki łącznej L929, zachodzących pod wpływem utrwalenia aldehydem glutarowym oraz w wyniku działania laurylosiarczanu sodu (SDS).
Najlepsze zrozumienie procesów biochemicznych zachodzących pod wpływem różnych czynników (np. leków) niosą badania prowadzone na żywych komórkach, co najbardziej odpowiada rzeczywistym stanom fizjologicznym. Jednakże nie zawsze jest to możliwe ze względu na krótki czas życia oraz szybko zachodzące procesy biochemiczne komórek podczas prowadzenia analiz. Z tego powodu badacze powszechnie wykorzystują substancje mające na celu utrwalenie materiału, czyli zatrzymanie procesów enzymatycznych (np. autolizy) czy rozkładu bakteryjnego, prowadzących do zmiany składu biochemicznego. Istotne jest jednakże aby w badaniach prowadzonych na komórkach utrwalonych uwzględnić wpływ substancji utrwalającej.
Na Rysunku 1 przedstawiono przykładowe wyniki obrazowania ramanowskiego komórek L929 poddanych działaniu utrwalacza w porównaniu do kontroli (komórka „żywa”). W komórkach traktowanych aldehydem glutarowym zaobserwowano zwiększenie powierzchni jądra komórkowego wraz ze spadkiem intensywności pasma pochodzącego od drgań grup fosforanowych DNA (785 cm-1), świadczących o zmniejszeniu gęstości upakowania chromatyny.9 Efekt utrwalenia widoczny jest również w spadku intensywności pasma ramanowskiego pochodzącego od cytochromu (750 cm-1), co wynika z jego utlenienia, a także w zmianie stosunków pasm 1659 oraz 1147 cm-1, sugerując zmiany w stopniu nienasycenia lipidów.6 Na widmach komórek utrwalonych pojawia się także dodatkowe pasmo położone przy 1033 cm-1. Wskazuje ono na zmiany konformacyjne w obrębie pierścienia aromatycznego fenyloalaniny, które zachodzą na skutek interakcji aldehydu glutarowego z białkami błony komórkowej.9
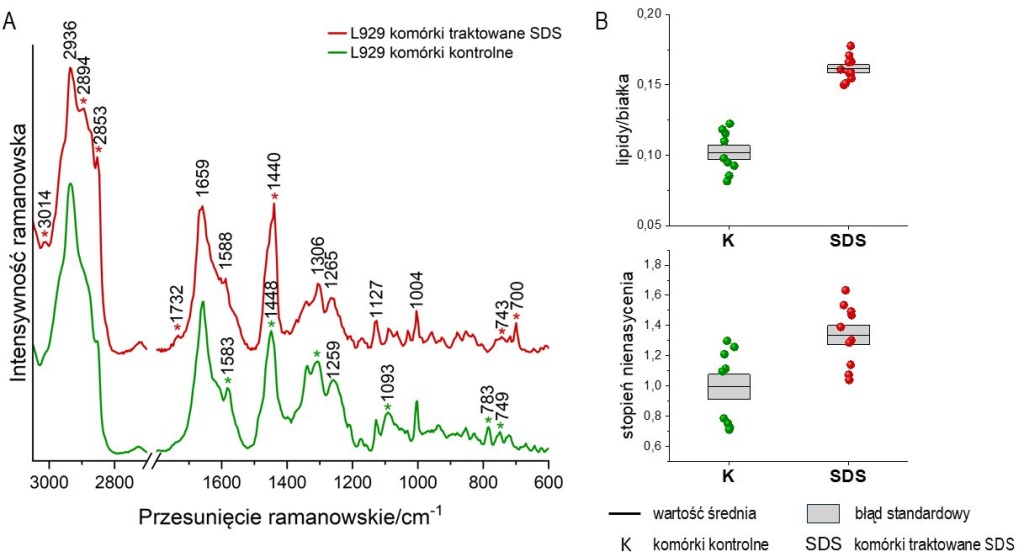
Celem kolejnego eksperymentu była detekcja zmian biochemicznych zachodzących podczas śmierci komórkowej (apoptozy). Nieutrwalone komórki L929 poddano działaniu SDS, środka używanego powszechnie jako detergent. Zastosowano wysokie stężenie tego związku, co zgodnie z oczekiwaniem doprowadziło do apoptozy. Wśród obserwowanych zmian biochemicznych potwierdzających ten proces należy wyróżnić spadek intensywności sygnału od kwasów nukleinowych (783 cm-1), świadczący o rozpadzie jądra komórkowego, pojawienie się cholesterolu oraz jego estrów w cytoplazmie (700 oraz 1732 cm-1), utlenienie cytochromu (spadek intensywności pasm 749 oraz 1583 cm-1), wzrost zawartości lipidów w cytoplazmie (2853 cm-1) oraz zmiany w drugorzędowej strukturze białek (Rysunek 2).1,9 Dzięki zastosowaniu analizy chemometrycznej, polegającej na policzeniu intensywności integralnej odpowiednich pasm (ich pola powierzchni), uzyskano pół-ilościową informację o wzroście stosunku lipidów do białek oraz stopnia nienasycenia kropli lipidowych w komórkach L929 traktowanych SDS.
Podsumowując, mikroskopia ramanowska może dostarczyć wielu cennych informacji o biochemicznych zmianach zachodzących w komórkach z podziałem na poszczególne struktury. Mogą mieć one kluczowe znaczenie w zrozumieniu bądź potwierdzeniu działania różnych związków na stan funkcjonalny komórki, jak również pomóc wyjaśnić podstawowe mechanizmy związane z powstawaniem i rozwojem stanów patologicznych.
Bibliografia
- Dybas, J. et al. Raman spectroscopy as a sensitive probe of soft tissue composition – Imaging of cross-sections of various organs vs. single spectra of tissue homogenates. TrAC – Trends Anal. Chem. 85, 117–127 (2016).
- Malek, K. Vibrational Spectroscopy. From theory to practise. (PWN, 2016).
- Butler, H. J. et al. Using Raman spectroscopy to characterize biological materials. Nat. Protoc. 11, 664–687 (2016).
- Szczesny‐Malysiak, E., Bartkowiak, A. & Dybas, J. Label‐free tracking of cytochrome C oxidation state in live cells by resonance Raman imaging. FEBS Lett. (2024) doi:10.1002/1873-3468.14905.
- Czamara, K. et al. Raman spectroscopy of lipids: A review. J. Raman Spectrosc. 46, 4–20 (2015).
- Okada, M. et al. Label-free Raman observation of cytochrome c dynamics during apoptosis. Proc. Natl. Acad. Sci. U. S. A. 109, 28–32 (2012).
- Perez-Guaita, D. et al. Parasites under the Spotlight: Applications of Vibrational Spectroscopy to Malaria Research. Chem. Rev. 118, 5330–5358 (2018).
- Desroches, J. et al. A new method using Raman spectroscopy for in vivo targeted brain cancer tissue biopsy. Sci. Rep. 8, 1792 (2018).
- Bik, E., Dorosz, A., Mateuszuk, L., Baranska, M. & Majzner, K. Fixed versus live endothelial cells: The effect of glutaraldehyde fixation manifested by characteristic bands on the Raman spectra of cells. Spectrochim. Acta Part A Mol. Biomol. Spectrosc. 240, 118460 (2020).
Po więcej informacji odwiedź profil Jagiellońskiego Centrum Innowacji na Platformie Współpracy LSOS.

Materiały do powyższego artykułu zostały dostarczone przez Jagiellońskie Centrum Innowacji.