Ilościowa reakcja łańcuchowa polimerazy w czasie rzeczywistym (ang. real time quantitative polymerase chain reaction, qPCR) to metoda analityczna szeroko stosowana zarówno w badaniach naukowych jak i diagnostyce.
W metodzie tej poziom ekspresji badanych genów normalizowany jest względem genu/genów referencyjnych. Aby gen referencyjny był prawidłowym wzorcem, poziom jego ekspresji powinien być stały niezależnie od czynników środowiskowych jakim podlega komórka lub jej stanu fizjologicznego.
Istotnym etapem metody jest właściwy wybór genów referencyjnych. Ich arbitralny dobór np. jedynie na podstawie literatury, skutkuje błędnym wyznaczeniem poziomu ekspresji genów badanych, a co za tym idzie błędną interpretacją wyników. Z kolei stosowanie jedynie ogólnie dostępnych programów komputerowych do wyboru referencji, np. geNorm, NormFinder czy BestKeeper prowadzi do uzyskania różnych wyników, co związane jest z ograniczeniami jakie te algorytmy ze sobą niosą.
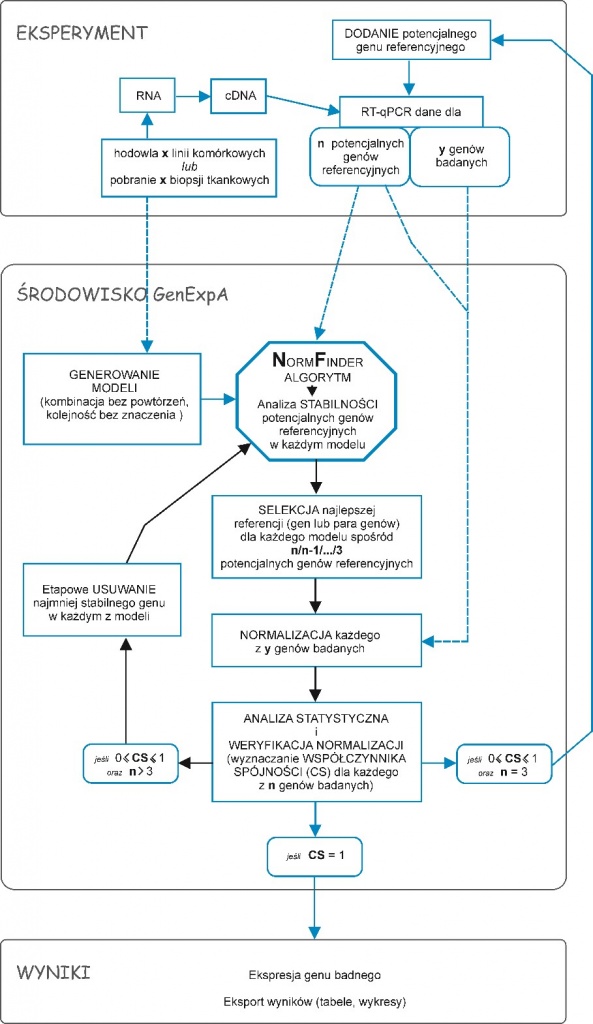
Próbując rozwiązać ten problem zespół naukowy z Wydziału Biologii Uniwersytetu Jagiellońskiego w składzie dr hab. Dorota Hoja-Łukowicz, prof. UJ oraz dr Marcelina Janik, opracował program komputerowy o nazwie GenExpA do analizy danych i wizualizacji wyników z metody RT-qPCR oparty o nowatorską metodę uzyskiwania ulepszonej referencji (Rys. 1).
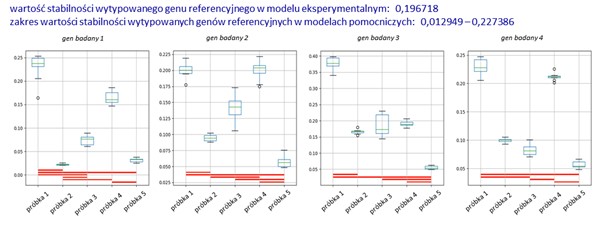
Program generuje podmodele − modele pomocnicze − z próbek wchodzących w skład modelu eksperymentalnego, przy czy najmniejsza liczba próbek w podmodelu to dwie. Następnie, przy użyciu zaimplementowanego algorytmu NormFinder, w każdym z tych modeli (model eksperymentalny oraz modele pomocnicze) typowany jest najlepszy gen lub para genów referencyjnych, które stanowią następnie podstawę do przeprowadzenia walidacji spójności wyników. Obliczane jest zatem RQ dla genów badanych i w ramach każdego z modeli prowadzone są analizy pokazujące istotne statystycznie różnice w ekspresji badanego genu w obrębie danego modelu. Kolejnym etapem jest porównanie spójności otrzymanych wyników/wartości i ich statystycznej istotności dla danego genu badanego w obrębie wszystkich badanych modeli. Przygotowany program wyznacza tzw. coherence score (współczynnik spójności; CS) opisujący poziom spójności otrzymanych wyników. Osiągnięcie niezadowalającej wartości współczynnika spójności, poniżej akceptowalnej przez badacza (domyślnie poniżej wartości 1), prowadzi do kolejnego etapu analizy, tj. ponownego wyboru genów referencyjnych we wszystkich badanych modelach, ale odbywa się to po uprzednim usunięciu z listy potencjalnych genów referencyjnych tego, który na poprzednim etapie uzyskał najsłabszy poziom stabilności w danym modelu (czyli np. z grupy wyjściowych n genów referencyjnych usuwany jest ten o najsłabszym wyniku stabilności i właściwy gen referencyjny jest wybierany spośród pozostałych n-1 potencjalnych genów referencyjnych). Dalej analiza prowadzona jest analogicznie jak poprzednio i zostaje zakończona przypisaniem współczynnika spójności dla genu badanego. Tym razem wartość współczynnika spójności jest obliczana na podstawie porównania wyników dla danego genu badanego w obu przeprowadzonych do tej pory etapach analizy (porównanie wyników obliczonych we wszystkich modelach na podstawie genu referencyjnego wybranego spośród n i spośród n-1 potencjalnych genów referencyjnych). Przy powtarzającym się braku wymaganego poziomu spójności analiza powtarzana jest kolejny raz po usunięciu we wszystkich modelach kolejnego potencjalnego genu referencyjnego o najsłabszym wyniku stabilności, tym razem spośród n-1 potencjalnych genów referencyjnych. Na każdym z etapów usuwania genu o najsłabszej stabilności, narzędzie generuje pliki informujące o otrzymanych wynikach analiz, przedstawionych w formie tabel oraz ostatecznie w formie wykresów pudełkowych (ang. box-plot), z naniesionymi na wykres informacjami o istotności statystycznej wyników (Rys. 2).
Rozwiązanie łączy w sobie funkcjonalności programów wyznaczających najlepsze geny referencyjne oraz programów typu arkuszy kalkulacyjnych, pozwalających na analizę poziomu ekspresji genu badanego, analizę statystyczną otrzymanych wyników i ich interpretację graficzną. Istotnym elementem rozwiązania jest zaimplementowanie określonego schematu analizy danych RT-qPCR (usuwanie genów o najsłabszej stabilności z następującą po tym ponowną analizą danych surowych) oraz wprowadzenie wartości współczynnika spójności określającego rzetelność/wiarygodność przeprowadzonej analizy. Usprawnieniem całości procesu jest także automatyczne generowanie wykresów pudełkowych prezentujących otrzymane wyniki analizy, włącznie z zaznaczonymi na wykresie istotnościami statystycznymi pomiędzy badanymi elementami danego modelu. Program umożliwia jednoczesną, niezależną analizę wielu genów badanych.
Opracowane narzędzie informatyczne umożliwia:
- Selekcję referencji, o ulepszonej wartości stabilności, z wyjściowej puli potencjalnych genów referencyjnych;
- Określenie rzeczywistego poziomu ekspresji genów badanych;
- Prawidłową interpretację biologiczną wyników na podstawie kompleksowej analizy danych surowych otrzymanych w reakcji RT-qPCR dla wieloelementowych modeli eksperymentalnych (duże grupy badawcze złożone z linii komórkowych, próbek pobranych od pacjentów lub zwierząt).
Obecnie Centrum Transferu Technologii CITTRU UJ poszukuje podmiotów zainteresowanych współpracą przy wdrażaniu wynalazku.
Program testowy można znaleźć na stronie www pod linkiem: www.sciencemarket.pl
Program GenExpA opracowany został z funduszy grantowych Narodowego Centrum Nauki, Polska (projekt badawczy nr 2016/21/B/NZ3/00348) oraz współfinansowany ze środków Programu Inkubator Innowacyjności 4.0 „Wsparcie zarządzania badaniami naukowymi i komercjalizacja wyników prac B+R w jednostkach naukowych i przedsiębiorstwach”, działanie 4.4 POIR 2014-2020.